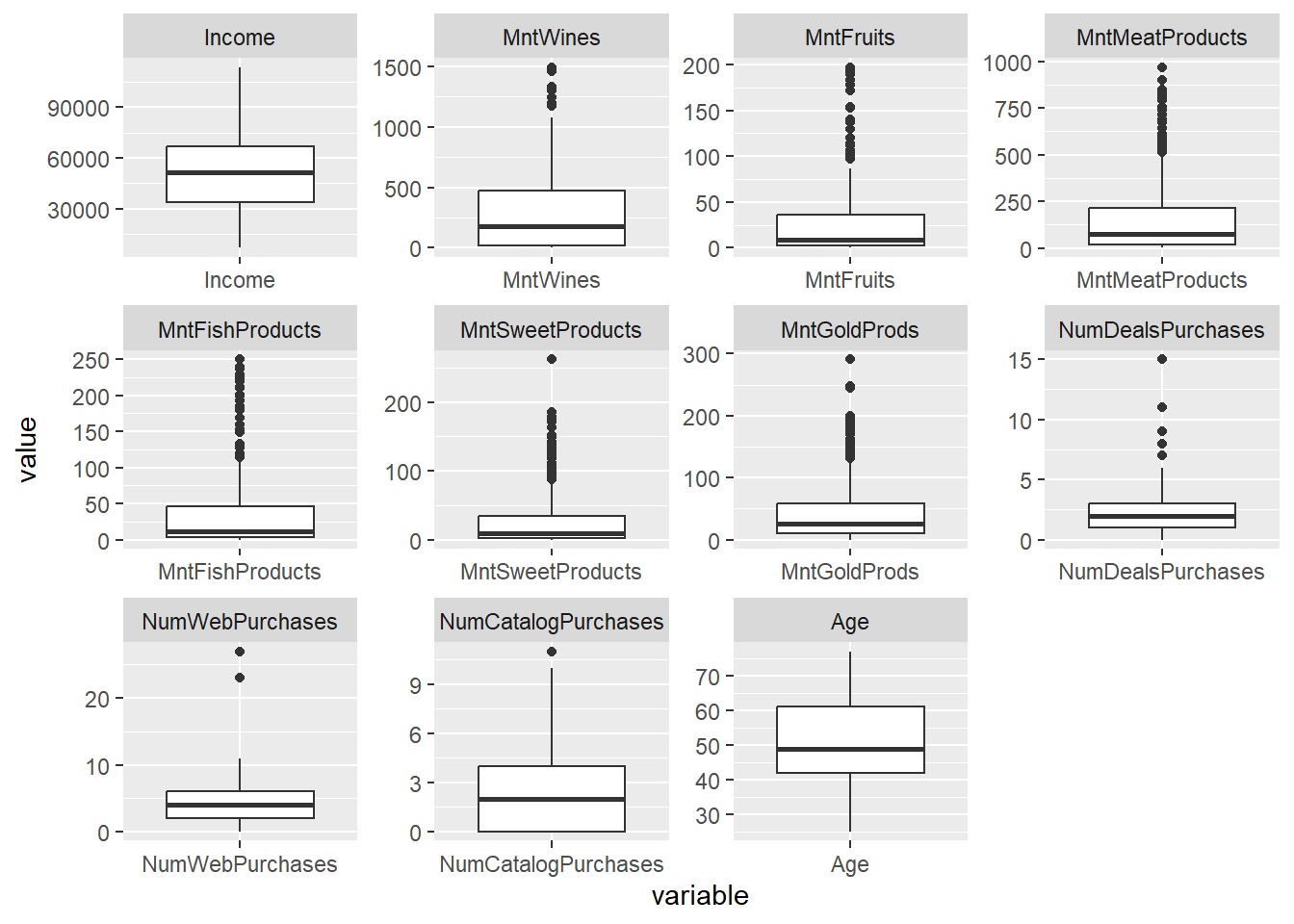
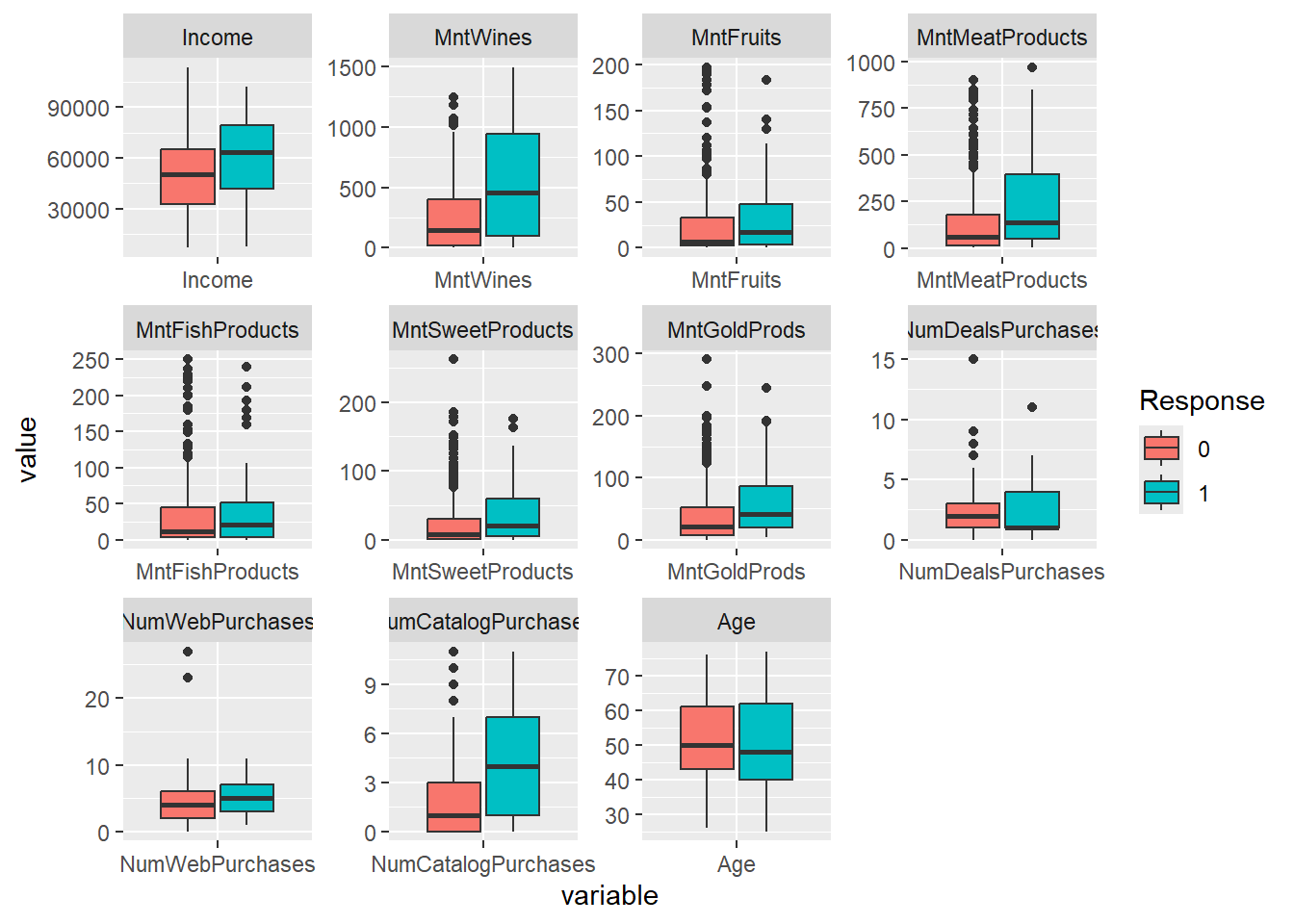
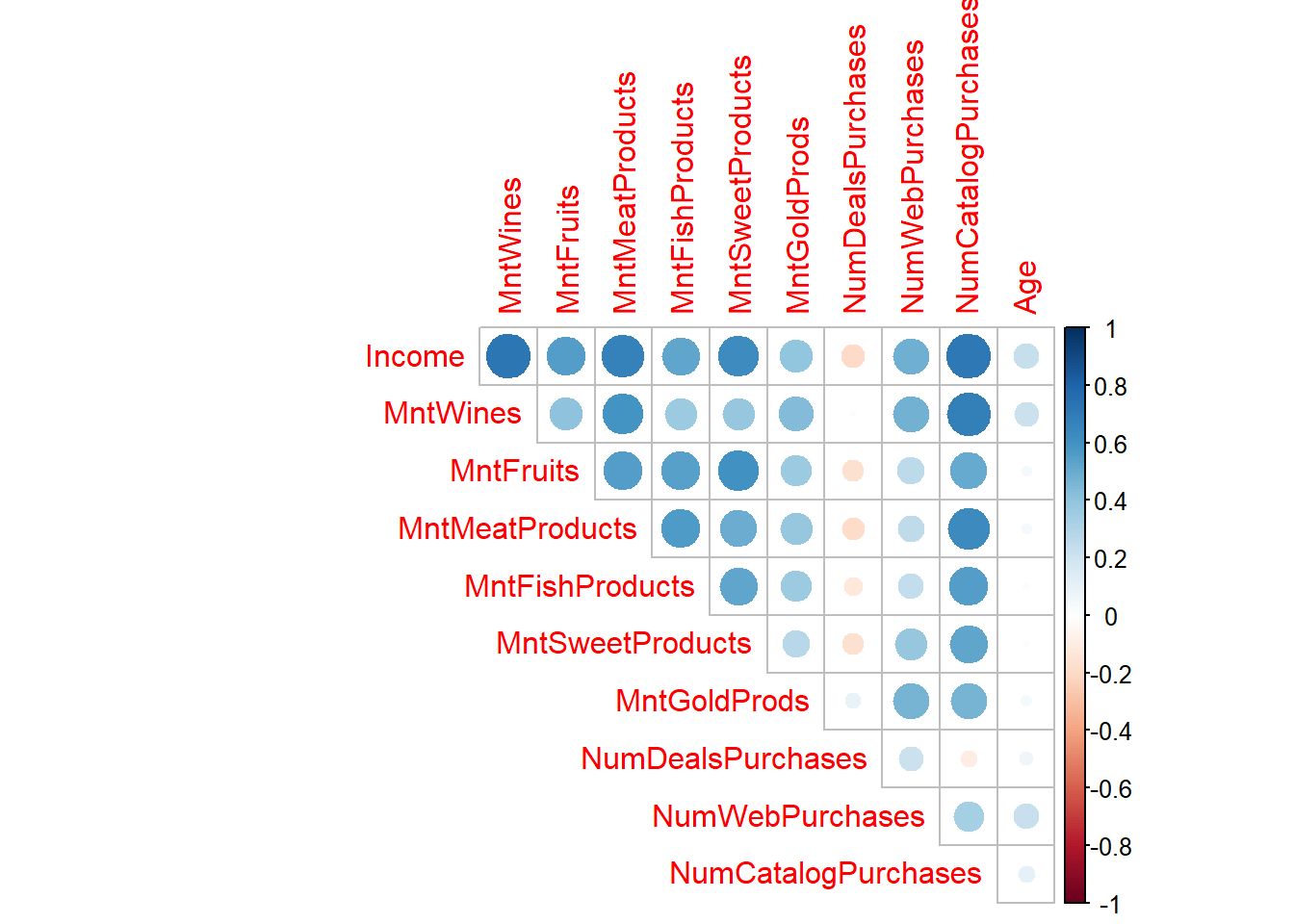
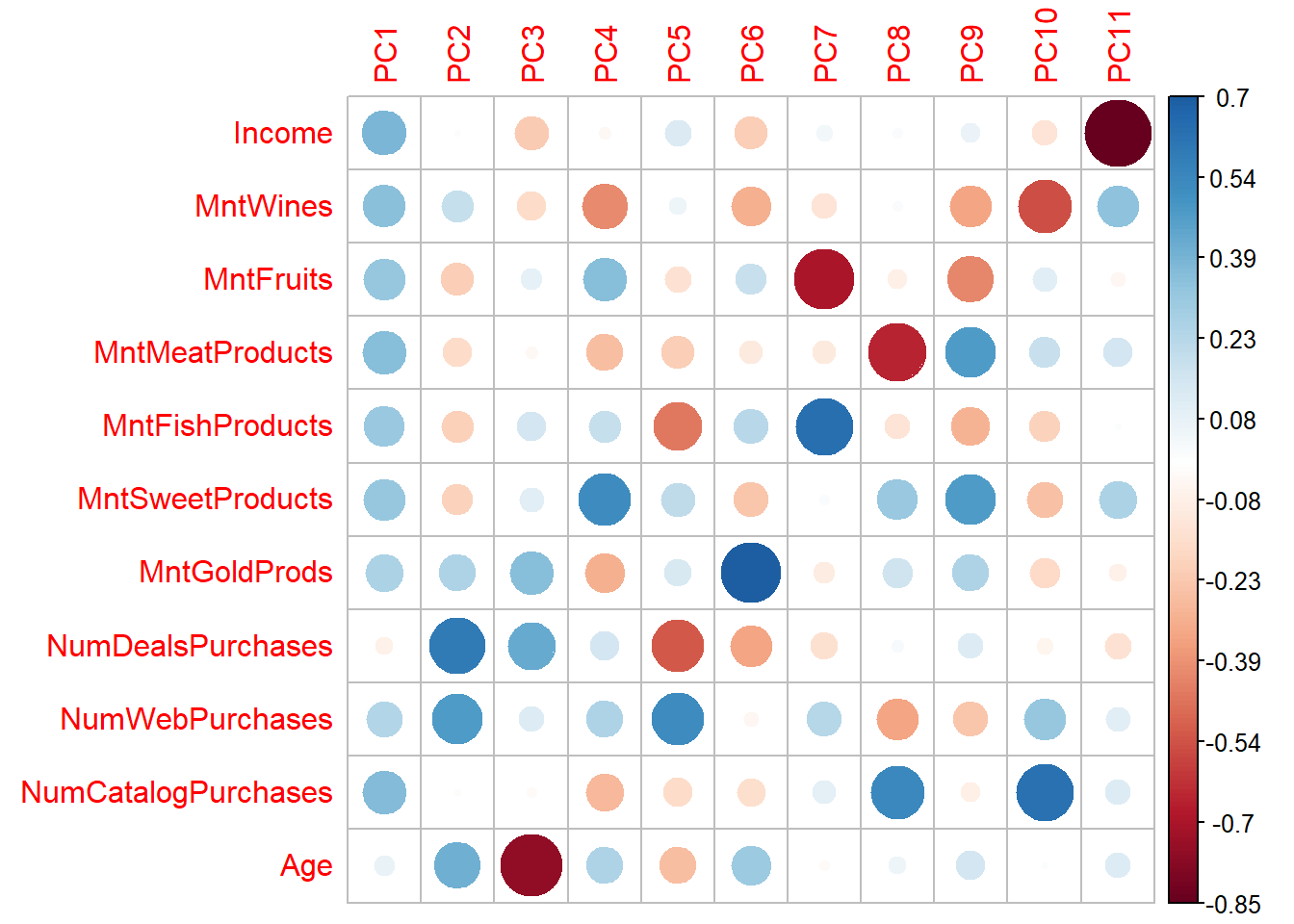
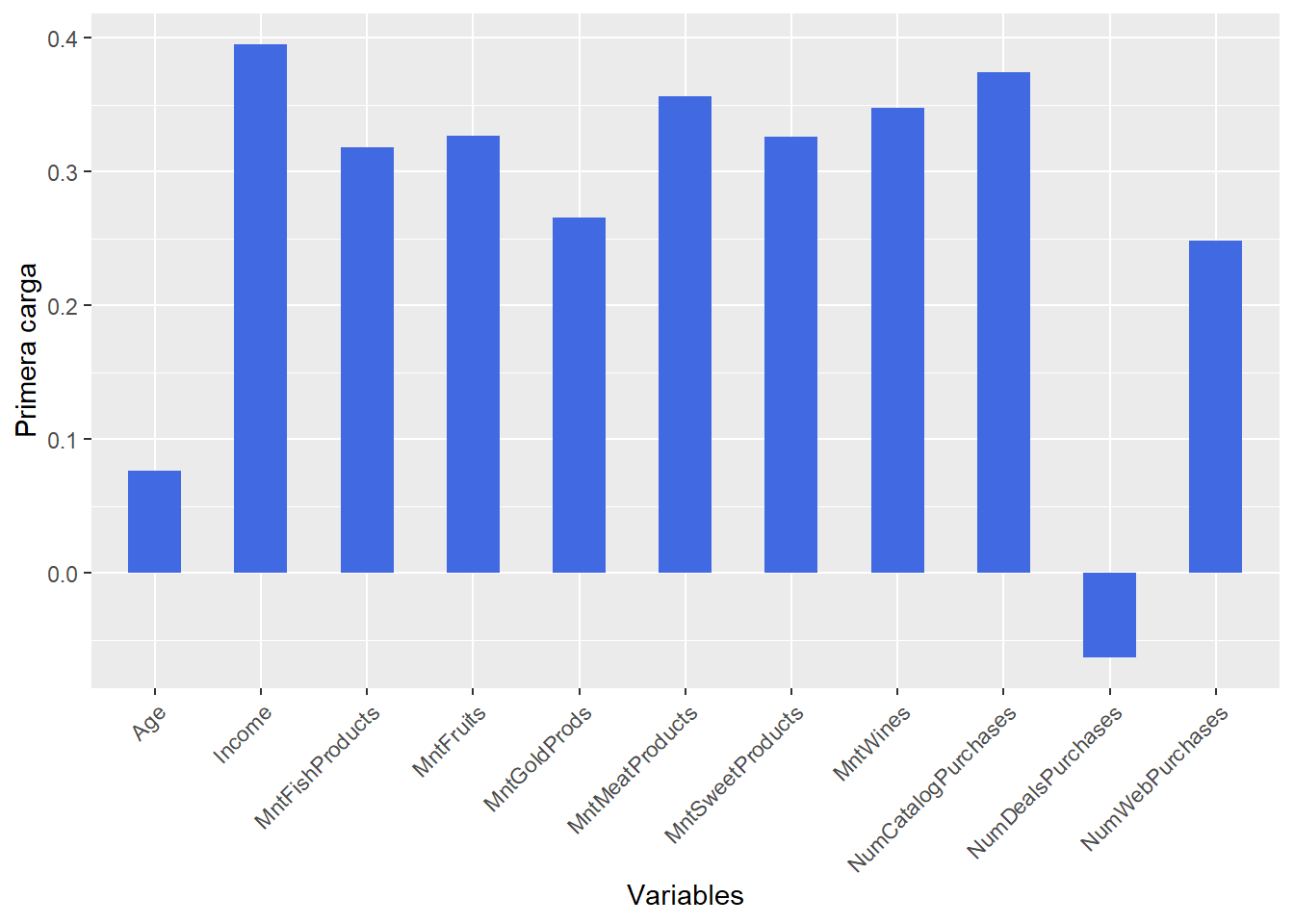
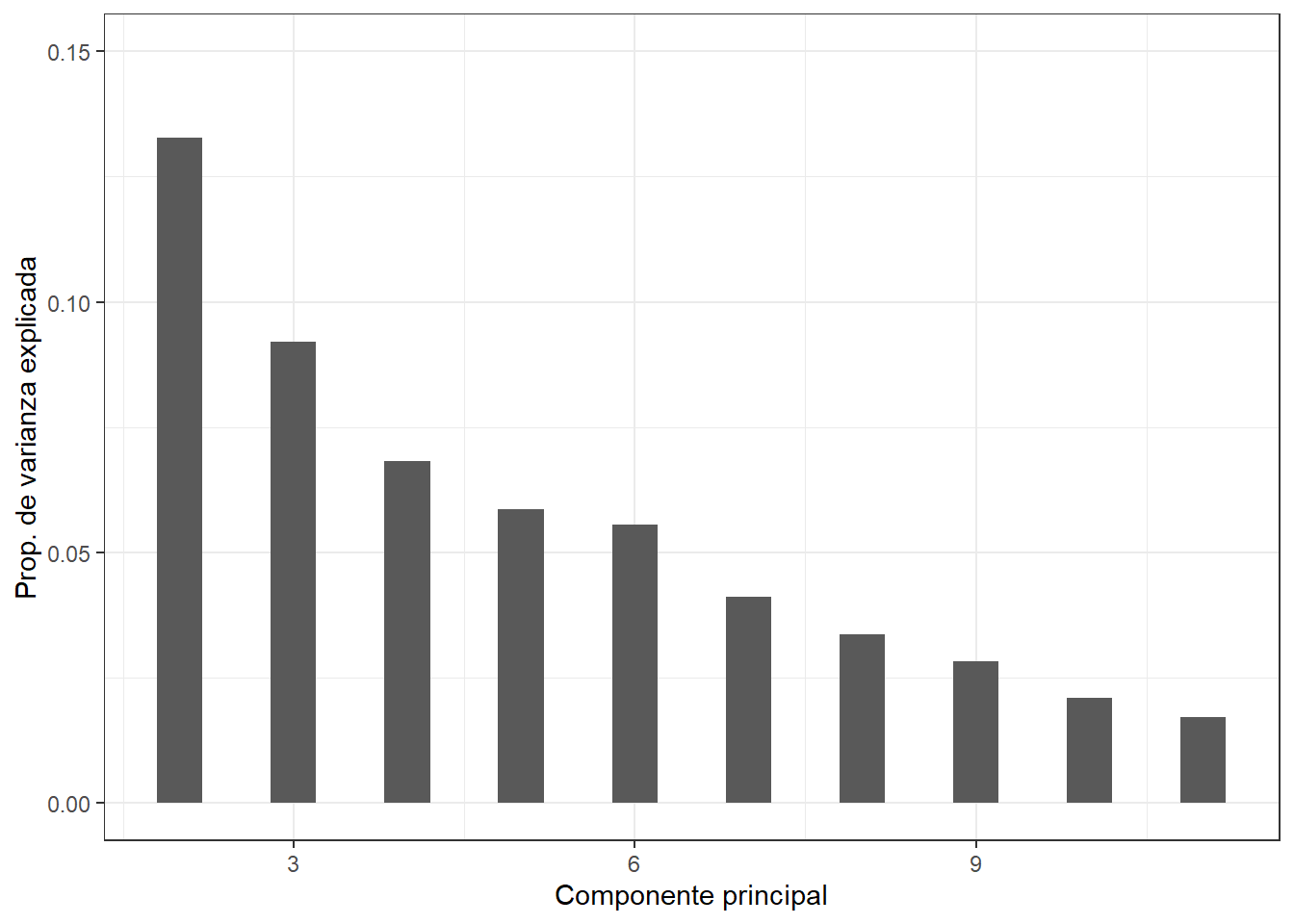
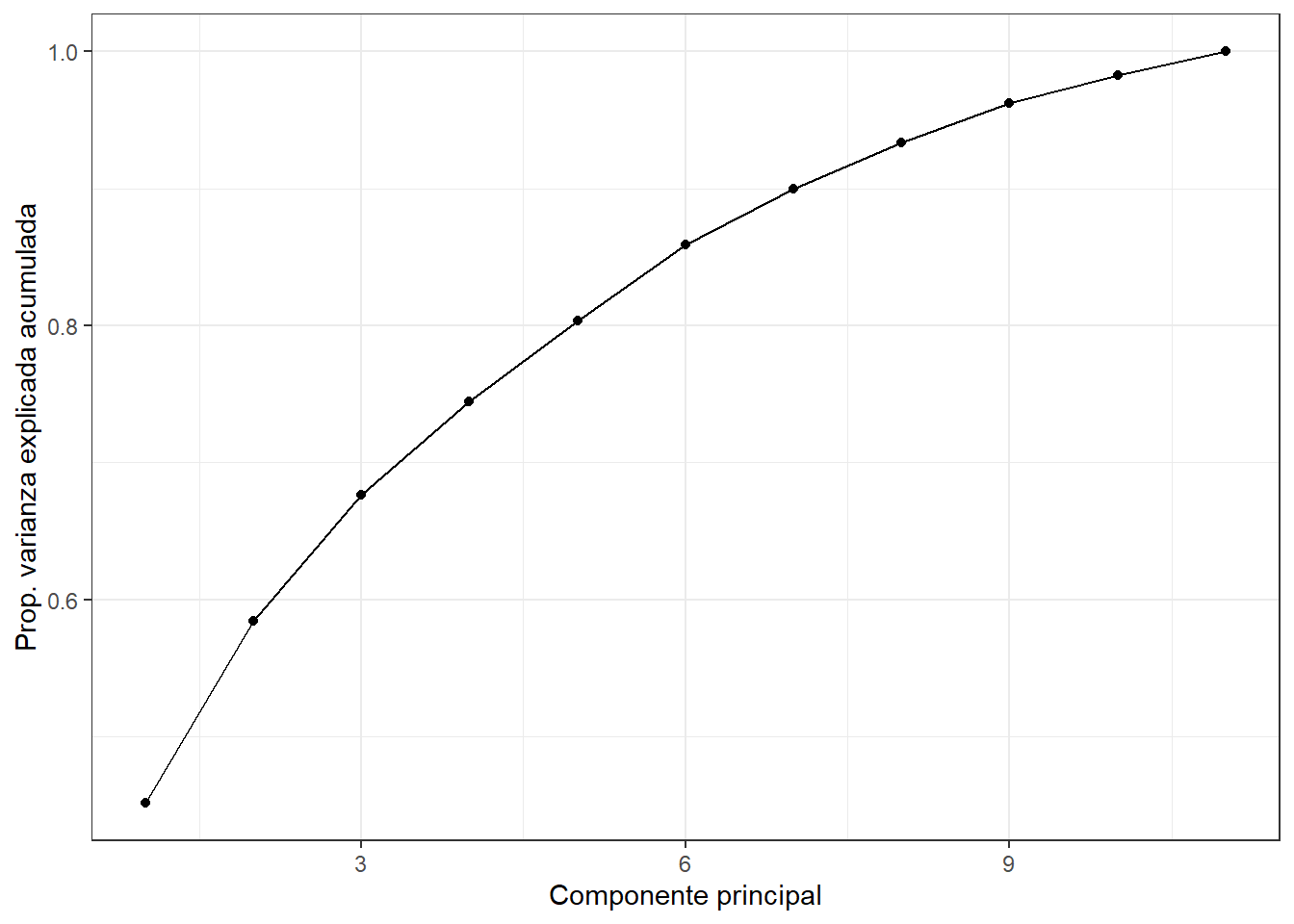
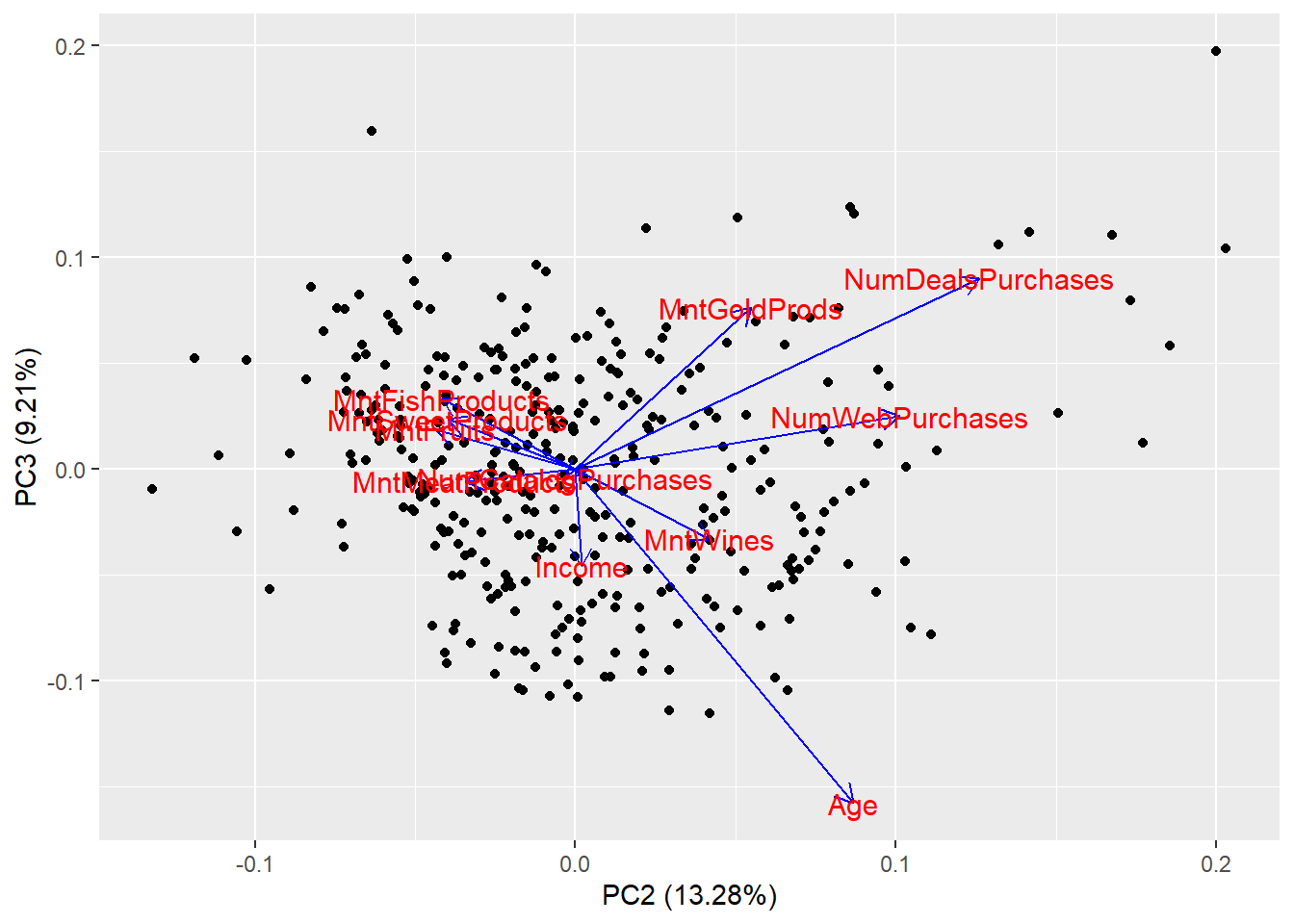
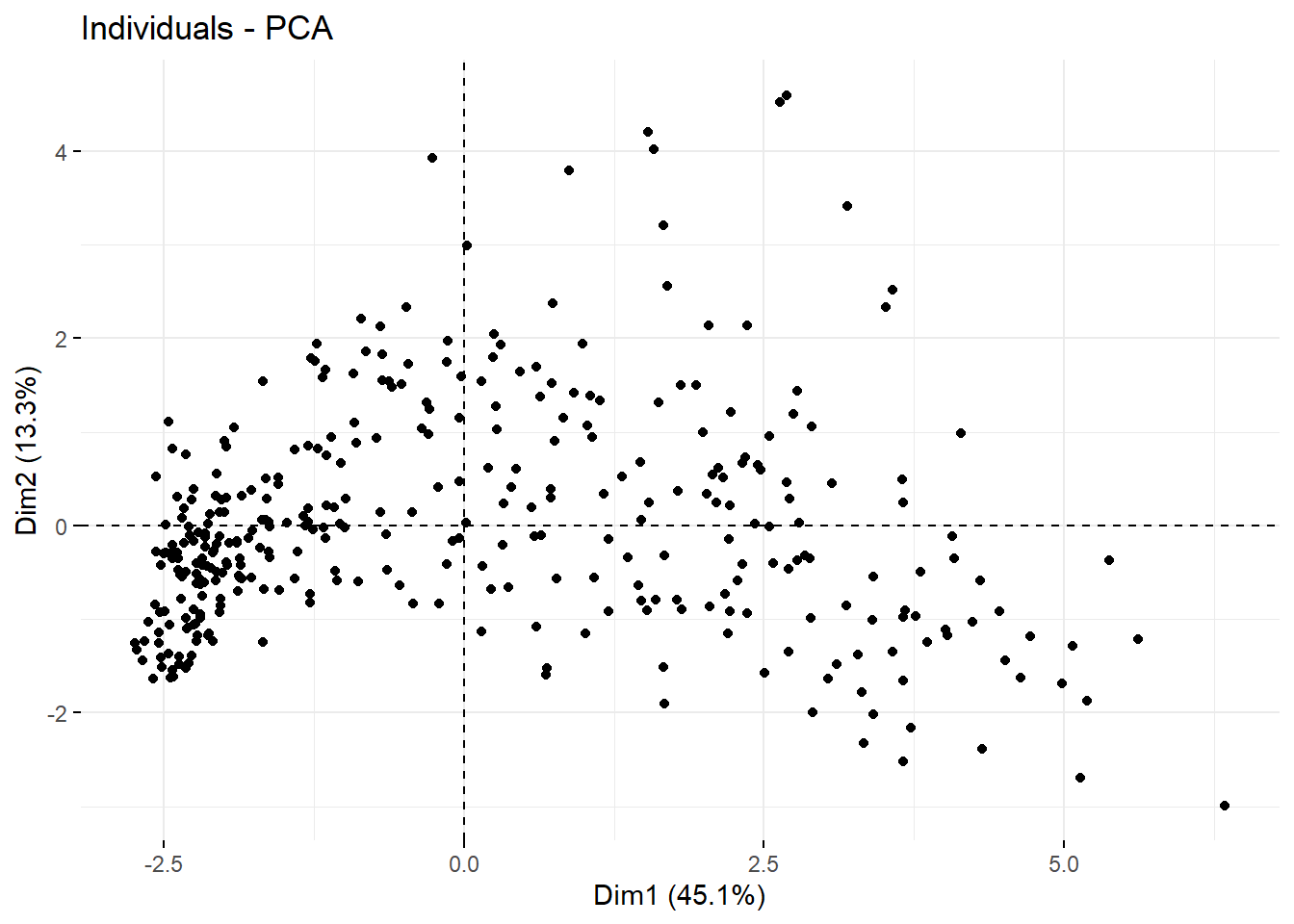
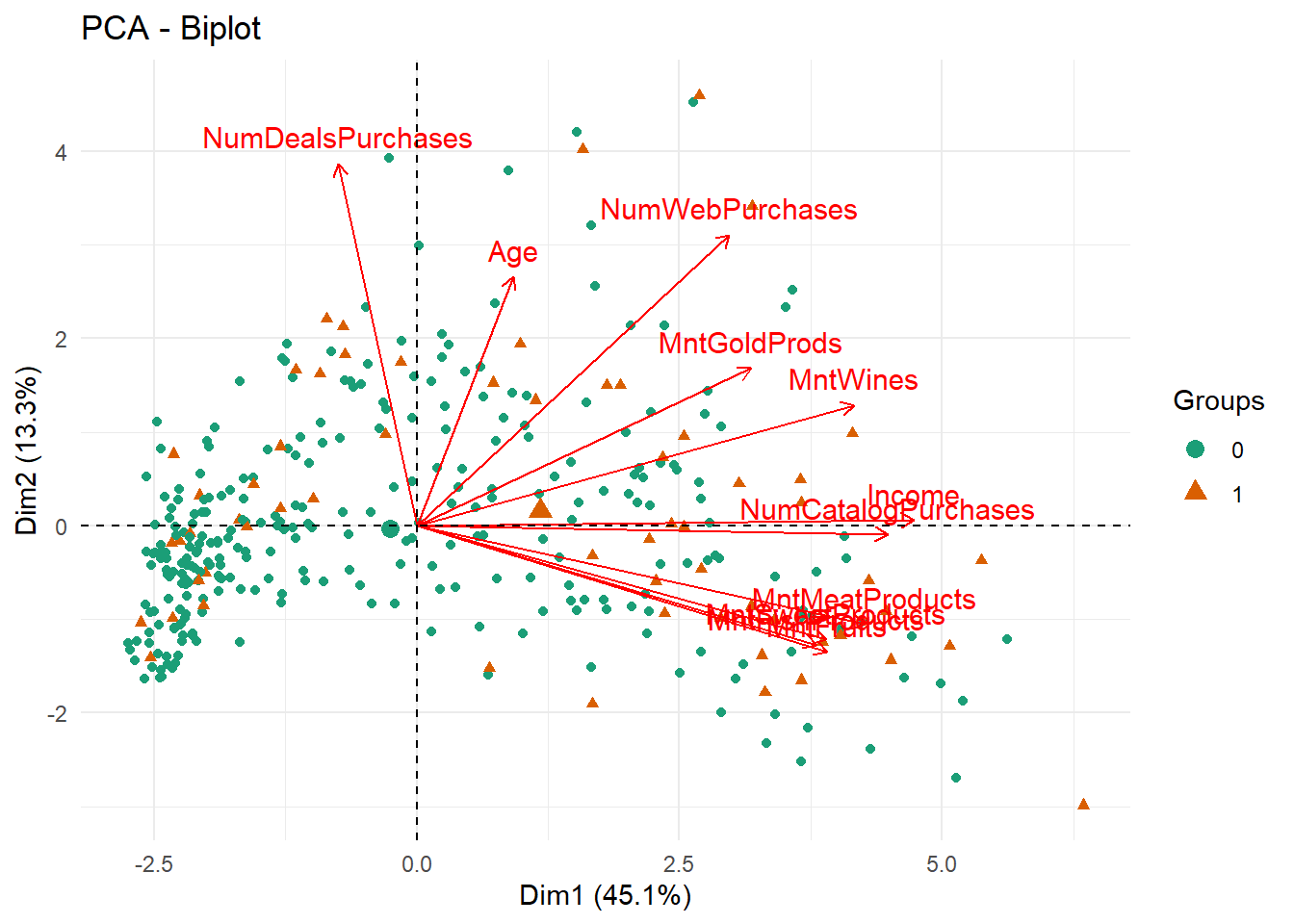
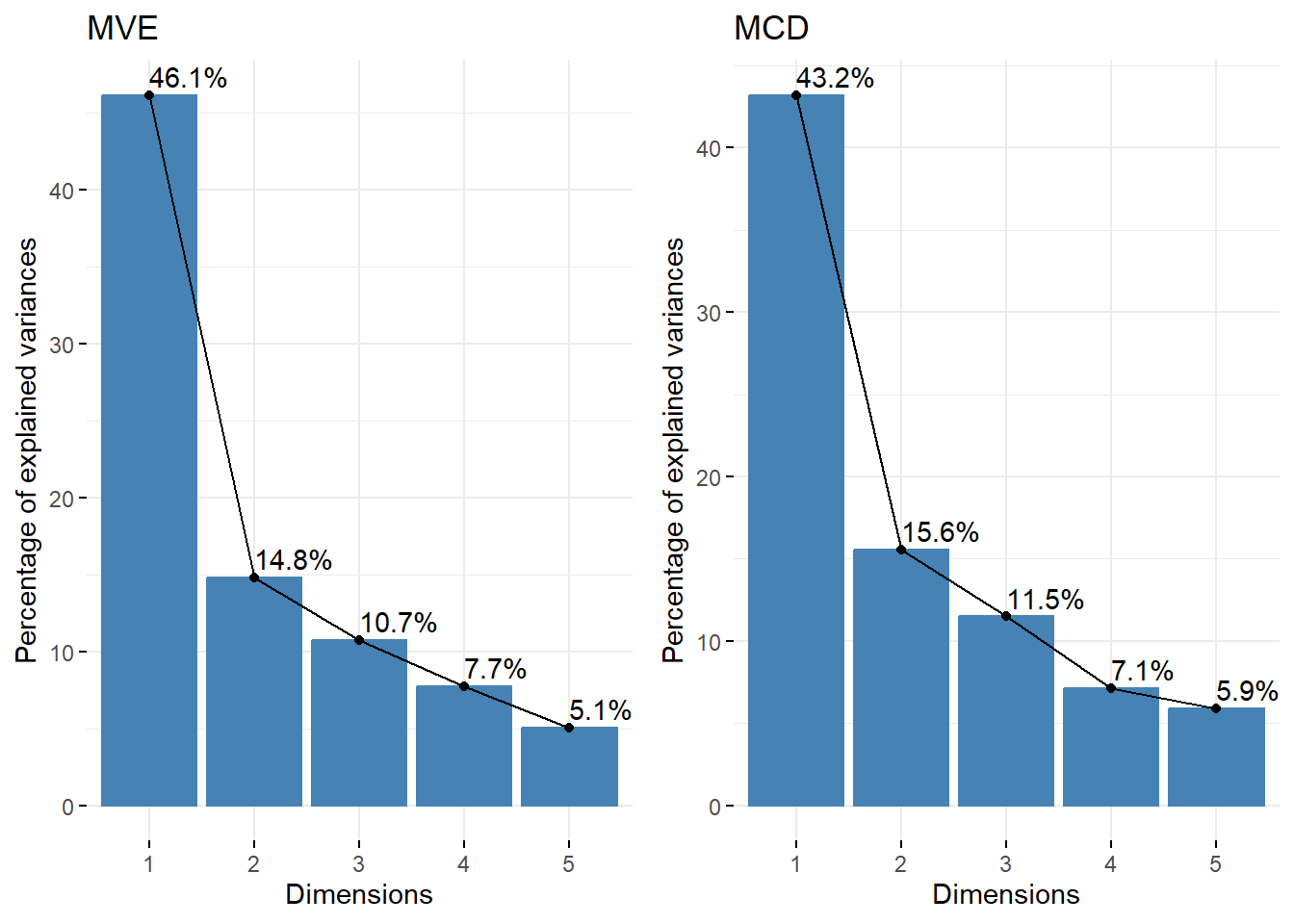
Rows: 2,205
Columns: 39
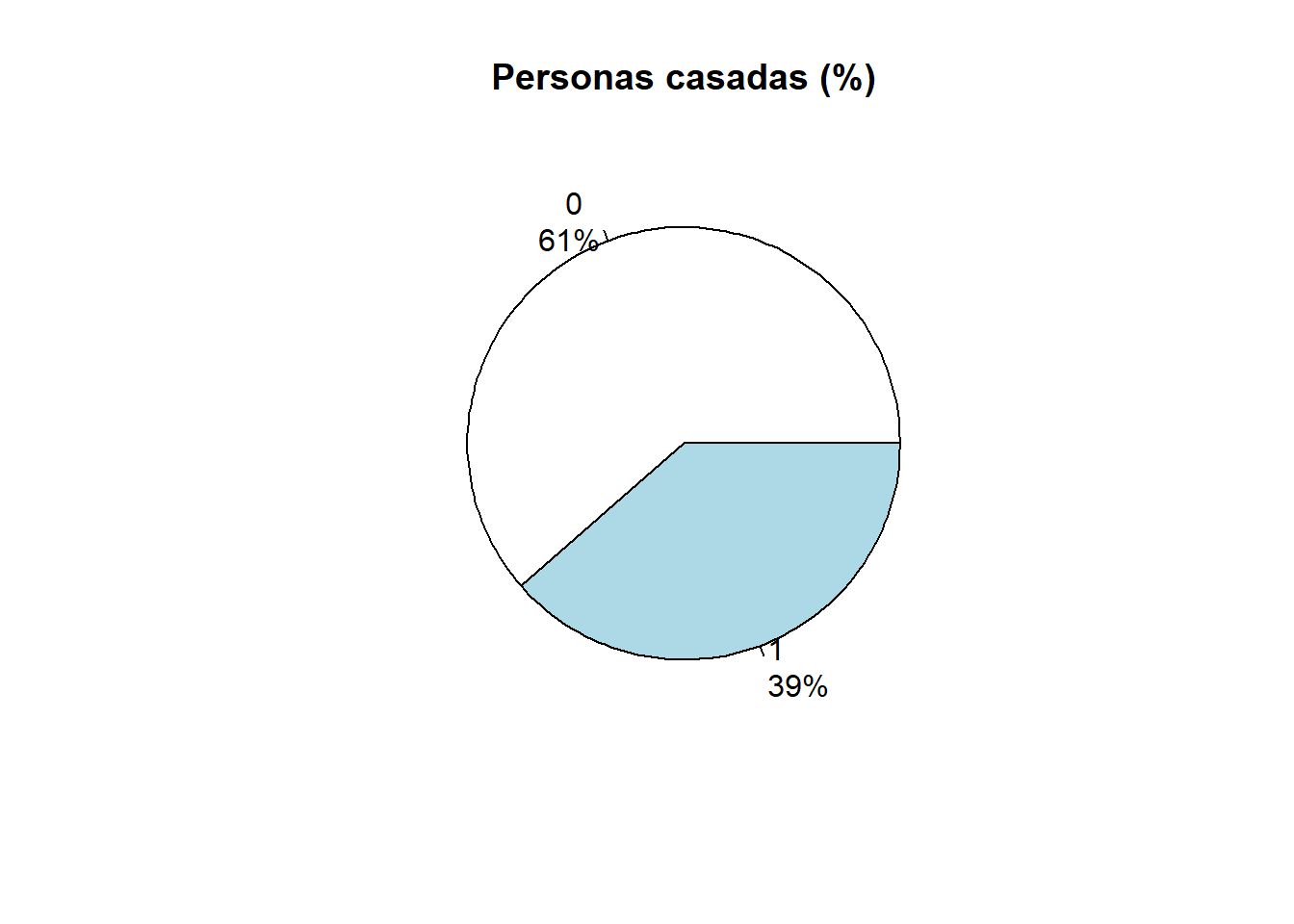
$ Income <dbl> 58138, 46344, 71613, 26646, 58293, 62513, 55635, …
$ Kidhome <int> 0, 1, 0, 1, 1, 0, 0, 1, 1, 1, 0, 0, 1, 0, 0, 1, 0…
$ Teenhome <int> 0, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 1, 0, 0, 1, 0…
$ Recency <int> 58, 38, 26, 26, 94, 16, 34, 32, 19, 68, 59, 82, 5…
$ MntWines <int> 635, 11, 426, 11, 173, 520, 235, 76, 14, 28, 6, 1…
$ MntFruits <int> 88, 1, 49, 4, 43, 42, 65, 10, 0, 0, 16, 61, 2, 14…
$ MntMeatProducts <int> 546, 6, 127, 20, 118, 98, 164, 56, 24, 6, 11, 480…
$ MntFishProducts <int> 172, 2, 111, 10, 46, 0, 50, 3, 3, 1, 11, 225, 3, …
$ MntSweetProducts <int> 88, 1, 21, 3, 27, 42, 49, 1, 3, 1, 1, 112, 5, 1, …
$ MntGoldProds <int> 88, 6, 42, 5, 15, 14, 27, 23, 2, 13, 16, 30, 14, …
$ NumDealsPurchases <int> 3, 2, 1, 2, 5, 2, 4, 2, 1, 1, 1, 1, 3, 1, 1, 3, 2…
$ NumWebPurchases <int> 8, 1, 8, 2, 5, 6, 7, 4, 3, 1, 2, 3, 6, 1, 7, 3, 4…
$ NumCatalogPurchases <int> 10, 1, 2, 0, 3, 4, 3, 0, 0, 0, 0, 4, 1, 0, 6, 0, …
$ NumStorePurchases <int> 4, 2, 10, 4, 6, 10, 7, 4, 2, 0, 3, 8, 5, 3, 12, 3…
$ NumWebVisitsMonth <int> 7, 5, 4, 6, 5, 6, 6, 8, 9, 20, 8, 2, 6, 8, 3, 8, …
$ AcceptedCmp3 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0…
$ AcceptedCmp4 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ AcceptedCmp5 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0…
$ AcceptedCmp1 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0…
$ AcceptedCmp2 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ Complain <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ Z_CostContact <int> 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3…
$ Z_Revenue <int> 11, 11, 11, 11, 11, 11, 11, 11, 11, 11, 11, 11, 1…
$ Response <int> 1, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0…
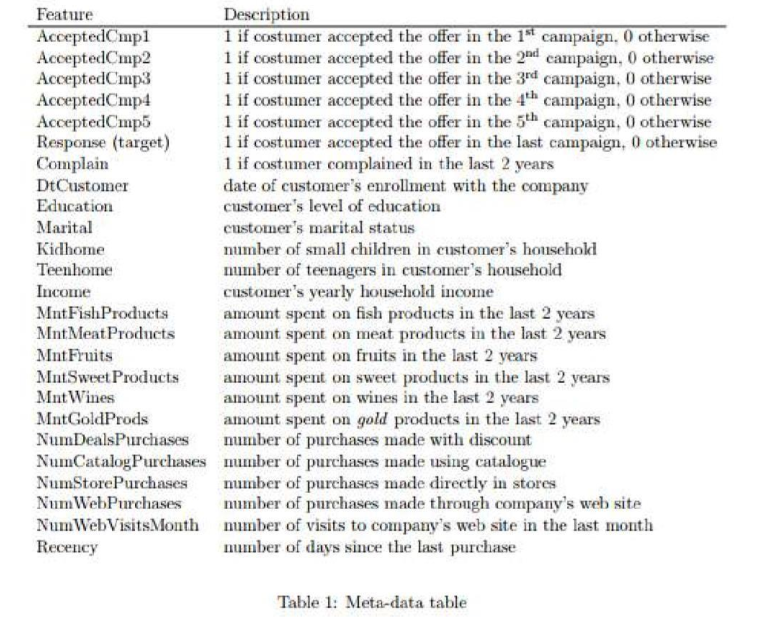
$ Age <int> 63, 66, 55, 36, 39, 53, 49, 35, 46, 70, 44, 61, 6…
$ Customer_Days <int> 2822, 2272, 2471, 2298, 2320, 2452, 2752, 2576, 2…
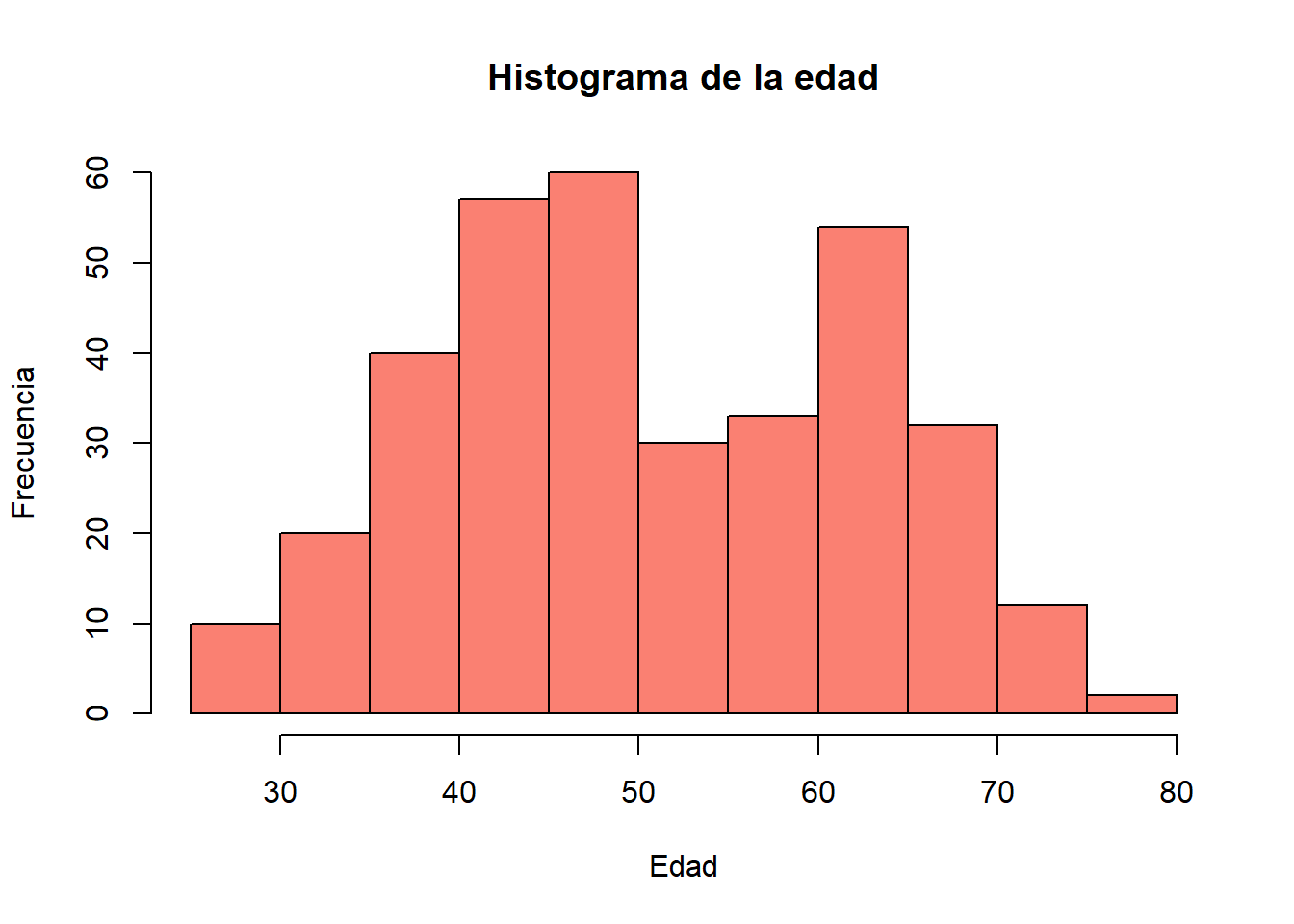
$ marital_Divorced <int> 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 1, 1, 0, 0, 0, 0…
$ marital_Married <int> 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 1, 0, 0, 1, 0, 1, 0…
$ marital_Single <int> 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0…
$ marital_Together <int> 0, 0, 1, 1, 0, 1, 0, 0, 1, 1, 0, 0, 0, 0, 0, 0, 1…
$ marital_Widow <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ education_2n.Cycle <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ education_Basic <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0…
$ education_Graduation <int> 1, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0, 1, 0, 1, 1…
$ education_Master <int> 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0…
$ education_PhD <int> 0, 0, 0, 0, 1, 0, 0, 1, 1, 1, 0, 0, 0, 0, 1, 0, 0…
$ MntTotal <int> 1529, 21, 734, 48, 407, 702, 563, 146, 44, 36, 45…
$ MntRegularProds <int> 1441, 15, 692, 43, 392, 688, 536, 123, 42, 23, 29…
$ AcceptedCmpOverall <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 2, 0, 0…